Un estudio demuestra que la Inteligencia Artificial puede descifrar la función de proteínas desconocidas
Un estudio demuestra que la Inteligencia Artificial puede descifrar la función de proteínas desconocidas
Es el primer estudio que demuestra que estas herramientas permiten clasificar las funciones previamente desconocidas con un gran nivel de detalle.
Este trabajo de colaboración entre dos centros del CSIC (CABD e IBE) permite identificar genes y explorar las proteínas que puedan ser de interés biomédico y biotecnológico, entre otras vías de investigación.
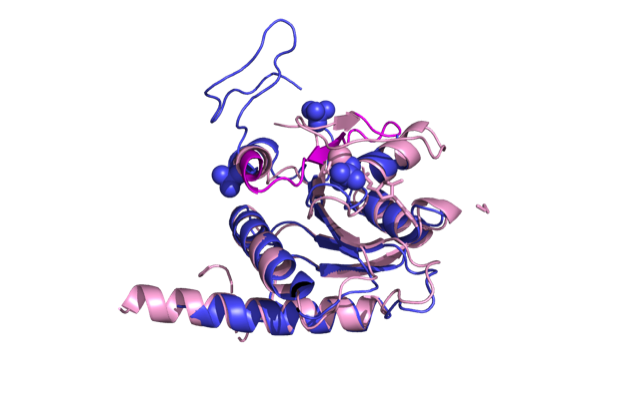
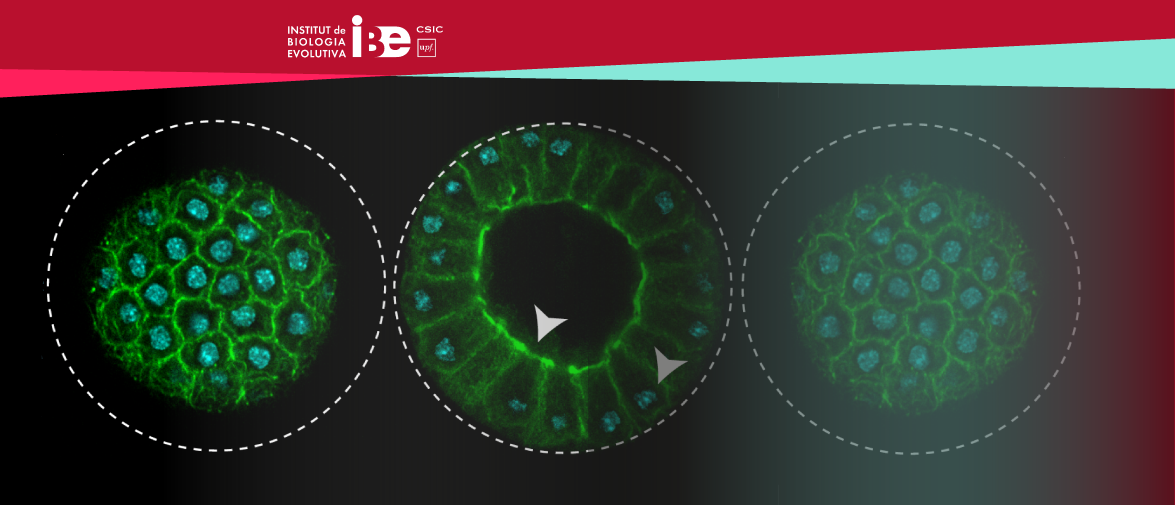
Un estudio del Centro Andaluz de Biología del Desarrollo (CABD-CSIC-UPO) junto al Instituto de Biología Evolutiva (IBE: CSIC-UPF) en Barcelona ha empleado técnicas avanzadas de inteligencia artificial para el análisis de proteínas. Gracias al uso de esta metodología, el equipo investigador ha conseguido demostrar que se puede identificar y describir lo que hacen las proteínas en detalle, incluso sin información previa. Este trabajo permite la aplicación masiva de estos métodos para entender las proteínas en organismos menos estudiados, identificar nuevas funciones de genes y explorar qué proteínas pueden ser de interés biomédico y biotecnológico con mucha más precisión que los métodos tradicionales.
En la naturaleza la información contenida en el ADN se transforma en proteínas, que son las que actúan en las células. En este proyecto, liderado por los investigadores del CABD, Ildefonso Cases y Ana M. Rojas junto a Rosa Fernández, del IBE, se han empleado dos métodos basados en el aprendizaje profundo (deep learning) para analizar proteínas en varios organismos modelo, como la levadura, el ratón o la mosca de la fruta. La exploración mostró que los modelos de lenguaje (Transformers) son más efectivos que las redes convolucionales, proporcionando información más precisa e informativa sobre las proteínas de las especies estudiadas. Además, los modelos de lenguaje pueden recuperar información funcional a partir de datos de ARN (el ARN es una molécula que lleva las instrucciones del ADN para fabricar proteínas en las células).
“Estamos en un momento crítico debido a la enorme cantidad de proyectos de secuenciación de organismos desconocidos que producen millones de secuencias, de las que no podemos predecir su función usando métodos tradicionales”, explica Ana Rojas (CABD). Este trabajo abre nuevas vías de investigación relacionadas con una mayor precisión en los modelos de análisis y clasificación de funciones de las proteínas.

Nuevas vías de investigación
Este nuevo estudio, publicado en la revista ‘Nuc Acids Red Genomics and Bioinformatics’, sienta las bases del uso de la inteligencia artificial en otras aplicaciones. “Estas herramientas de aprendizaje profundo permitirán abordar nuevos problemas en biología computacional. Estamos trabajando en la aplicación de estas técnicas para otros objetivos, como promotores a la carta, anotación de grupos de células en single-cell, o ingeniería de proteínas”.
Por su parte, la investigadora del IBE, Rosa Fernández, hace hincapié en que esta investigación es fundamental en el campo de la biodiversidad, donde cada día se publican nuevas secuencias de proteínas cuya función es desconocida, permitiendo abordar el problema de anotación del proteoma oscuro (Dark Proteome). “Para ello estamos usando estas herramientas en miles de transcriptomas del reino animal, trabajo que se encuentra en revisión. Cuanta más información tengamos de las funciones de secuencias nuevas, más rápido descifraremos los mecanismos moleculares de procesos biológicos que se dan en el ámbito de la biodiversidad y regeneración con potenciales aplicaciones biotecnológicas (industria alimentaria) y biomédicas (industria farmacéutica)”, concluye la investigadora.
Referencia del artículo:
Israel Barrios-Núñez, Gemma I Martínez-Redondo, Patricia Medina-Burgos, Ildefonso Cases, Rosa Fernández, Ana M Rojas, Decoding functional proteome information in model organisms using protein language models, NAR Genomics and Bioinformatics, Volume 6, Issue 3, September 2024, lqae078, https://doi.org/10.1093/nargab/lqae078