La biología sintética reinventa el desarrollo
La biología sintética reinventa el desarrollo
Un equipo de investigación liderado por investigadores del Instituto de Biología Evolutiva (IBE, CSIC-UPF) ha desarrollado con biología sintética un nuevo tipo de diseño genético capaz de reproducir algunos de los procesos clave que permiten crear estructuras en los sistemas naturales, desde los nidos de termitas hasta el desarrollo de los embriones. Este circuito sintético, basado en el modelo bacteriano E. coli, es capaz de inducir la formación de patrones espaciales regulares durante el crecimiento de la colonia bacteriana solo modificando genéticamente tres ingredientes esenciales de este organismo. El sistema sintético permite generar patrones inexistentes en la naturaleza y podría emplearse para diseñar otros sistemas (incluyendo células de mamífero) que presentan una organización espacial compleja y estudiar su formación y posibles alteraciones.
Richard Feynman, uno de los físicos más respetados del siglo XX, decía que “si no puedo crear algo es que no lo entiendo”. No en vano, muchos físicos y matemáticas han observado procesos biológicos fundamentales con la intención de identificar justamente los mínimos ingredientes que los pudieran generar. Un ejemplo lo encontramos en los patrones de la naturaleza observados por Alan Turing. El genial matemático inglés demostró en 1952 que era posible explicar cómo de un tejido totalmente homogéneo se podía crear un embrión complejo, y lo hizo con uno de los modelos matemáticos más elegantes y sencillos que se han escrito. Uno de los resultados de este tipo de modelos es que la simetría que muestra una célula o un tejido se puede “romper” bajo un conjunto de condiciones. Aun así, Turing no pudo poner a prueba sus ideas, y tuvieron que pasar más de 70 años antes de que los avances de la técnica en biología pudieran evaluarlas de forma contundente. ¿Se puede hacer realidad el sueño de Turing mediante la propuesta de Feynman? La ingeniería genética ha permitido demostrar que sí.
El equipo de investigadores ha desarrollado con biología sintética un mecanismo para generar patrones espaciales que se observan en animales más complejos.
Ahora, un equipo de investigación del Instituto de Biología Evolutiva (IBE), un centro mixto de la Universitat Pompeu Fabra (UPF) y del Consejo Superior de Investigaciones Científicas (CSIC), ha desarrollado un nuevo tipo de modelo y su implementación con biología sintética capaz de reproducir la ruptura de simetría observada en los embriones con la mínima cantidad de ingredientes posible.
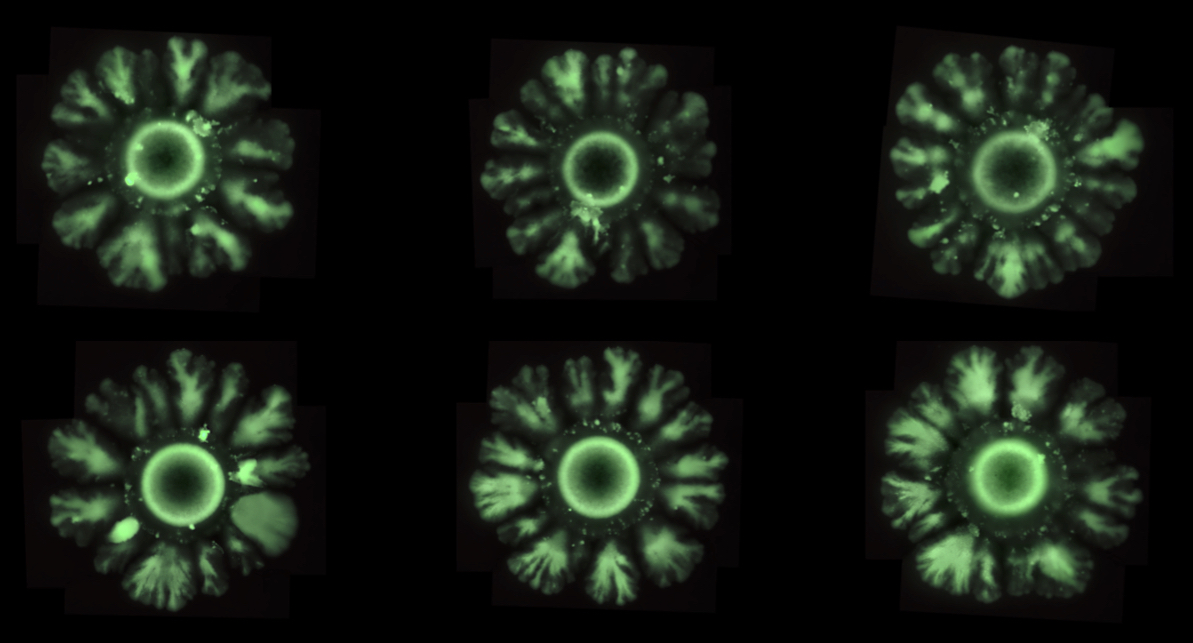
El equipo de investigación ha conseguido implementar con biología sintética (introduciendo en la bacteria E. coli piezas de genes propios de otros organismos) un mecanismo para generar patrones espaciales que se observan en animales más complejos, como Drosophila melanogaster (mosca de la fruta) o los humanos. En el estudio, el equipo ha observado cómo las cepas de E. coli modificadas, que normalmente crecen en colonias circulares (simétricas), lo hacen en forma de flor con pétalos a intervalos regulares, tal y como Turing predijo.
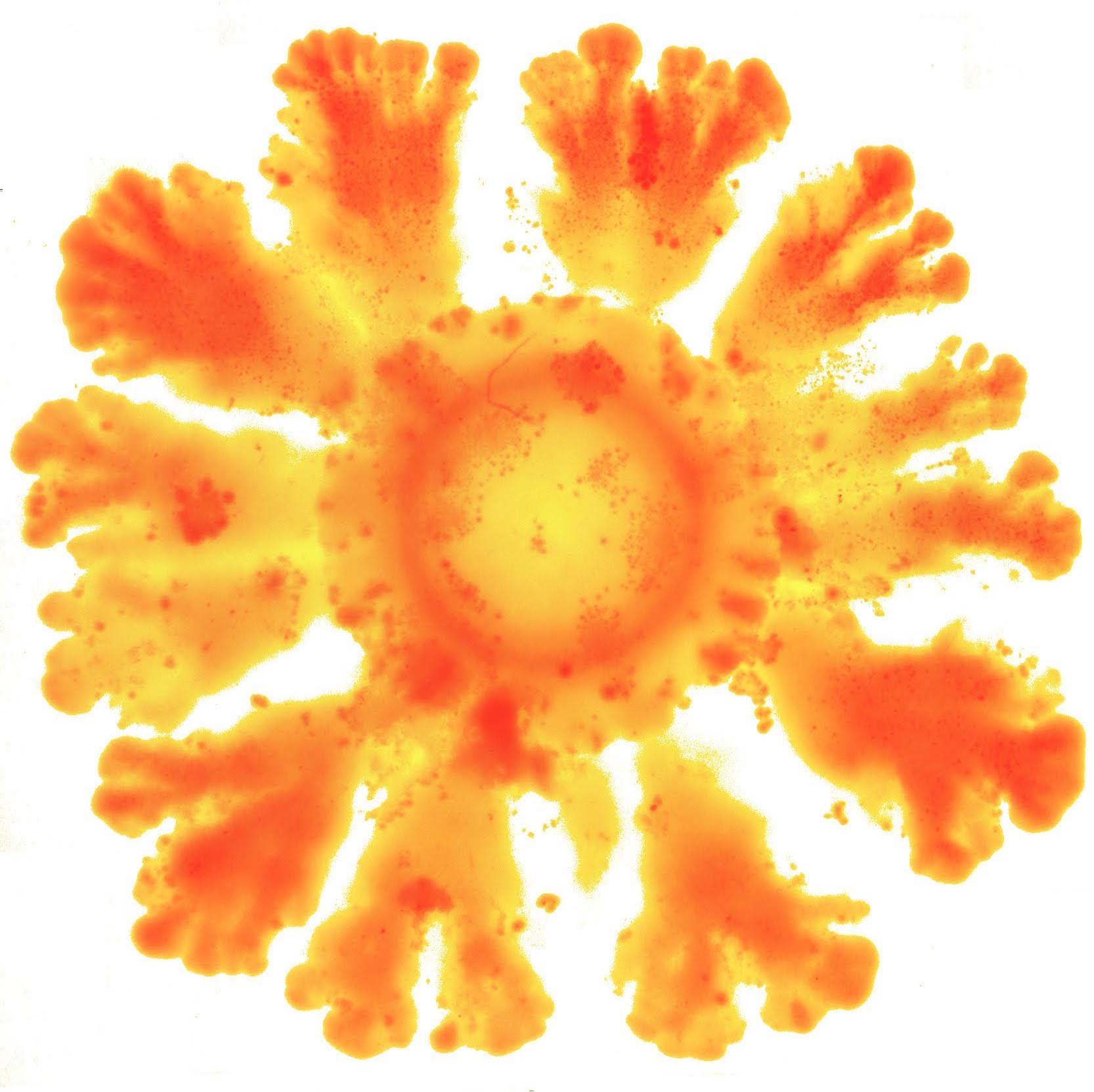
“Lo que queríamos era construir una ruptura de simetría que no se ve nunca en las colonias de E. coli, pero que se ve en patrones de animales, y a partir de aquí descubrir cuáles son los ingredientes indispensables que se necesitan para generar estos patrones” comenta Salva Duran-Nebreda, que realizó esta investigación durante su doctorado en el laboratorio de Sistemas Complejos y actualmente es investigador postdoctoral en el laboratorio de Evolución de la Tecnología del IBE.
Utilizando la nueva plataforma sintética, el equipo de investigación ha podido identificar los parámetros que modulan la aparición de patrones espaciales en E. coli. “Hemos visto que modulando tres ingredientes podemos inducir la ruptura de simetría. En esencia, hemos alterado la división celular, la adhesión entre células y la capacidad de comunicación a largas distancias (quorum sensing), es decir, percibir cuando hay una decisión colectiva”, comenta Duran-Nebreda.
El sistema sintético permite generar patrones inexistentes en la naturaleza y podría emplearse para diseñar otros sistemas que presentan una organización espacial compleja y estudiar su formación y posibles alteraciones.
Las observaciones hechas en el modelo E. coli podrían ser aplicadas a modelos animales más complejos o a los principios de diseño de las colonias de insectos. “Del mismo modo que los organoides u órganos en miniatura pueden ayudarnos a desarrollar terapias sin tener que usar modelos animales, este sistema sintético abre la puerta a la comprensión de un fenómeno tan universal como es el desarrollo embrionario en un sistema in vitro mucho más sencillo”, comenta Ricard Solé, investigador ICREA en el grupo de Sistemas Complejos del IBE y responsable de la investigación.
El modelo desarrollado en este estudio, el primero de su tipo, podría ser clave para entender algunos acontecimientos del desarrollo embrionario. “Tenemos que pensar en este sistema sintético como en una plataforma para aprender a diseñar diferentes mecanismos biológicos fundamentales que generan estructuras, como es el paso de un cigoto a la formación de un organismo completo. Además, este conocimiento en la frontera entre procesos mecánicos y biológicos podría ser muy útil para entender patologías del desarrollo”, concluye Duran-Nebreda.
Artículo de referencia: Duran-Nebreda S, Pla J, Vidiella B, Piñero J, Conde-Pueyo N, Solé R. Synthetic Lateral Inhibition in Periodic Pattern Forming Microbial Colonies. ACS Synth. Biol. 2021. https://doi.org/10.1021/acssynbio.0c00318
