Un árbol evolutivo más completo para los escarabajos
Un equipo de investigación del Instituto de Biología Evolutiva (IBE) ha combinado información taxonómica, genética, paleontológica y biogeográfica para deducir la historia evolutiva de la subfamilia de escarabajos Cryptocephalinae, la cuarta mayor entre los hiperdiversos Chrysomelidae, con más de 5.300 especies distribuidas por todo el mundo.
A través del análisis global de 167 especies de escarabajos de esta subfamilia, el equipo de investigación ha inferido el árbol filogenético más completo de este grupo megadiverso hasta la fecha.
El estudio demuestra la importancia de procesos globales, tanto tectónicos como climáticos, para explicar la diversidad y distribución de las líneas evolutivas que podemos reconocer hoy.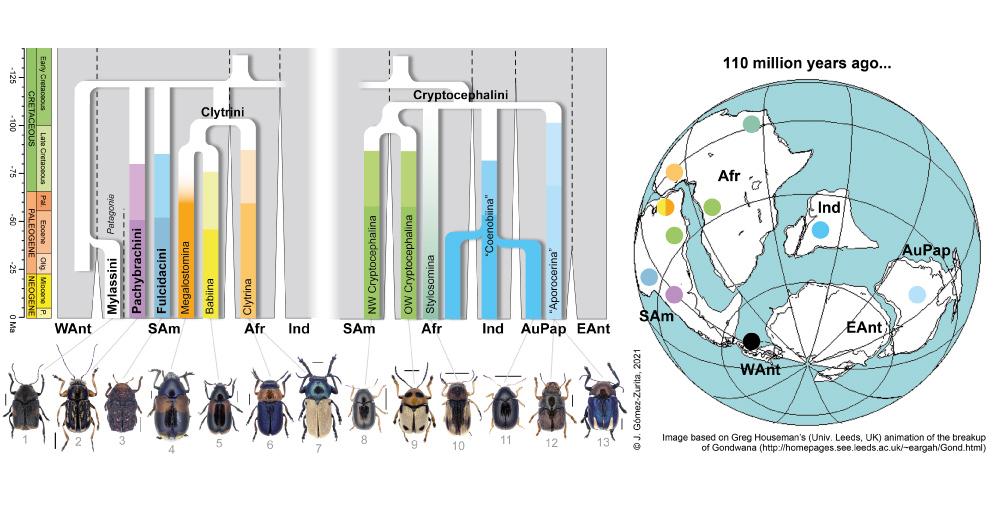
Los escarabajos juegan un papel esencial en los ecosistemas globales. Además, estos insectos se utilizan como modelos para el estudio de la evolución y como indicadores biológicos de la diversidad en los ecosistemas o de efectos sobre la biodiversidad por cambios globales. Sin embargo, la información genética y filogenética relativa a este numeroso grupo es limitada y sigue faltando un árbol evolutivo completo para este orden.
Ahora un estudio liderado por investigadores del Instituto de Biología Evolutiva (IBE), un centro mixto del Consejo Superior de Investigaciones Científicas (CSIC) y la Universidad Pompeu Fabra (UPF), ha inferido la historia evolutiva de la numerosa subfamilia de escarabajos Cryptocephalinae a través de información taxonómica, genética, biogeográfica y paleontológica. Para ello, han obtenido secuencias de ADN de 167 especies actuales de Cryptocephalinae de los cinco continentes.
La investigación liderada por Jesús Gómez-Zurita, incorporado recientemente con su equipo al Instituto Botánico de Barcelona (CSIC-Ajuntament de Barcelona), ha generado un árbol filogenético de estos escarabajos con más y mejores marcadores filogenéticos y diez veces más taxones que estudios anteriores. El árbol se ha calibrado basándose en el registro fósil y la edad conocida de una radiación insular, asignando fechas a ciertos hitos de la diversificación del grupo. El estudio ha sido publicado recientemente en la revista Zoologica Scripta.
El equipo analizó un extenso conjunto de datos genéticos en 167 especies de Cryptocephalinae, que representarían la mitad de los géneros de la subfamilia. “La investigación supone un paso adelante en el estudio de las relaciones evolutivas de estos escarabajos y un nivel de detalle hasta ahora desconocido sobre la estructura filogenética y la clasificación de este grupo”, comenta Jesús Gómez-Zurita, quien ha dirigido el estudio desde el laboratorio de Evolución de escarabajos herbívoros del IBE.
El análisis genético de 167 especies de la subfamilia de escarabajos Cryptocephalinae ha generado un árbol filogenético con más y mejores marcadores, así como diez veces más taxones que en estudios anteriores
El equipo de investigación ha empleado un reloj molecular calibrado utilizando información paleontológica y biogeográfica para situar el origen de esta subfamilia hace más de 116 millones de años en el megacontinente austral Gondwana, antes de su fragmentación. "Los diferentes linajes de Cryptocephalinae que existen posiblemente deben su origen al aislamiento resultante de la fragmentación de los principales territorios de Gondwana en el Cretácico Temprano", añade Gómez-Zurita.
El nuevo árbol filogenético, fruto de más de 20 años de muestreo y secuenciación genética de 190 especímenes en todo el mundo, "proporciona una herramienta fundamental para la clasificación natural de una porción pequeña pero significativa del Árbol de la Vida, para indagar los procesos que han generado esta enorme diversidad, y para dirigir futuros muestreos que permitan resolver dudas concretas sobre la evolución de estos organismos", concluye Gómez-Zurita.
Artículo de referencia: Gómez-Zurita, J. & Cardoso, A. 2021. Molecular systematics, higher-rank classification and Gondwanan origins of Cryptocephalinae leaf beetles. Zoologica Scripta, 50 (en prensa). DOI: 10.1111/zsc.12501
